"Wer bin ich und wenn ja wie viele?" lautet der Titel eines bekannten Buches von Richard David Precht. Bei dem größten Bakterium Deutschlands, Achromatium oxaliferum, kann man diese Frage mit aller wissenschaftlichen Ernsthaftigkeit stellen. Normalerweise tragen Bakterien als die einfachsten Lebewesen ihre genetische Information auf einem einzelnen Chromosom und evtl. einigen Plasmiden. Man kennt Ausnahmen davon, etwa bei großen Schwefelbakterien wie Thiomargarita nambiensis. Diese Bakterien sind bekannt als polyploid. Sie besitzen mehrere Chromosomen, bei denen es sich um identische Kopien handeln soll. Ein Forscherteam des Leibniz-Instituts für Gewässerökologie und Binnenfischerei (IGB) Berlin und des Instituts für Chemie und Biologie des Meeres (ICBM) der Universität Oldenburg hat nun das größte bekannte Süßwasserbakterium, Achromatium oxaliferum, näher untersucht und die Ergebnisse in der Fachzeitschrift Nature Communications publiziert.
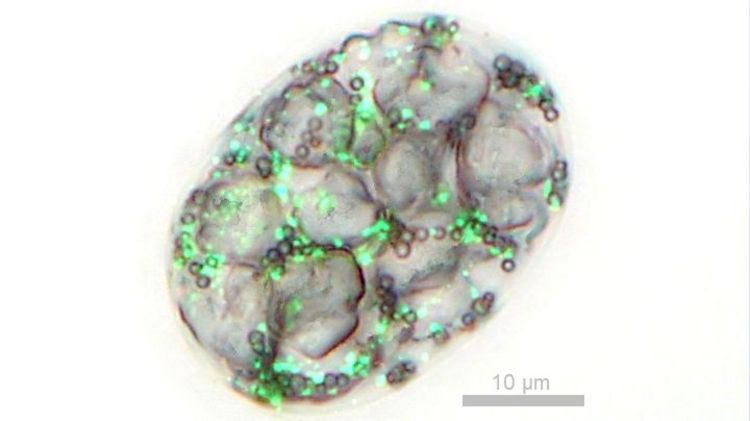
Dieses Bakterium, das im Labor noch nicht kultiviert werden konnte, wächst in den Sedimenten des Stechlinsees an der oxisch-anoxischen Grenzschicht. Einzelne Zellen sind so groß wie kleine Sandkörner, so dass man sie vor schwarzem Hintergrund mit dem bloßen Auge als helle Punkte sehen kann. Die schwefeloxidierenden Bakterien sind nicht nur angefüllt mit Calciumkristallen und Schwefeltöpfchen, sondern enthalten auch bis zu 300 Spots von DNA, die man mit geeigneten Farbstoffen unter dem Fluoreszenz-Mikroskop sichtbar machen kann (s. Abbildung).
Mit Hilfe von Metagenomik und Einzel-Zell-Genomanalyse konnten die Forscher zeigen, dass diese Spots nicht etwa identische DNA-Kopien enthalten. Stattdessen fanden sie eine Vielzahl verschiedener ribosomaler (rRNA) und Protein-kodierender Gene. Die sequenzspezifische Anfärbung von rRNA (mit Hilfe von Fluoreszenz-in-situ-Hybridisierung, FISH) zeigte, dass in verschiedenen Bereichen einzelner Zellen jeweils andere Gene exprimiert waren. Einzelne Achromatien, die morphologisch eine Zelle bilden und sich auch so teilen, beherbergen demnach verschiedene Phylotypen, wie man sie für eine ganze Population erwarten würde und bilden einen "bakteriellen Superorganismus".
Die Evolution dieses Phänomens ist ein spannendes Thema. Möglicherweise spielt Transposon-Mutagenese eine Rolle, da die Zellen eine sehr große Zahl von springenden Genen (Transposons) enthalten.
Zu diesem Thema erschien am 6. September 2017 ein Pressedienst, den Sie hier lesen können.
Weblinks: