Kontakt
- Leitung
Prof. Dr. Olaf R.P. Bininda-Emonds - Adresse
Carl von Ossietzky Universität Oldenburg
Fakultät V, Institut für Biologie und Umweltwissenschaften (IBU)
AG Systematik und Evolutionsbiologie
Carl von Ossietzky Str. 9-11
26129 Oldenburg
Raum: W04-2-217
Tel.: +49 (441) 798 3965
Fax: +49 (441) 798 193965
Email: olaf.bininda@uol.de - Wie Sie uns finden
Zufahrtswege zur Universität Oldenburg
Lageplan Campus Wechloy
(Math.-naturwiss. Standort)
Supertrees und Phyloinformatik
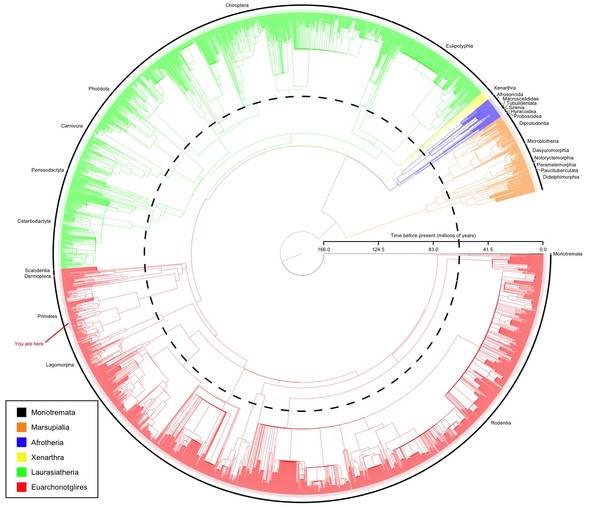
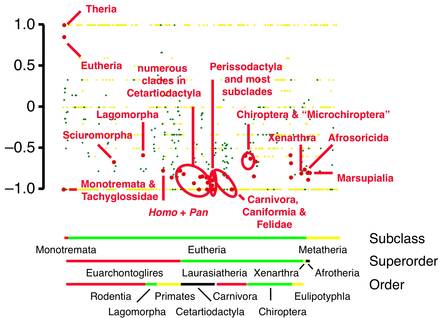
Die molekulare Revolution, kombiniert mit der Verfügbarkeit von Sequenzdaten in öffentlichen Datenbanken wie GenBank, Swiss-Prot, oder HOVERGEN, hat die Bandbreite phylogenetischer Rückschlüsse ungemein gesteigert. Nie zuvor waren so viele informative Daten erhältlich. Allerdings hat die Welle neuer Daten auch eine Welle neuer Fragen und Probleme mit sich gebracht: Wie können wir die Daten am besten analysieren um eine möglichst umfangreiche und stabile Stammesgeschichte zu erreichen?
Supertreekonstruktion
|
Phyloinformatik
|