Künstliche Intelligenz könnte die Suche nach einem Medikament gegen SARS-CoV-2 beschleunigen, sagt KI-Experte Oliver Kramer. Mit seinem Team hat er eine Methode entwickelt, um vielversprechende Ansätze zu identifizieren.
Ob Spracherkennung, Bildanalyse oder Online-Shopping: Künstliche Intelligenz (KI) ist bereits heute in zahlreichen Alltagsprodukten zu finden. Auch in der Medizin bescheinigen Fachleute der Zukunftstechnologie großes Potential. So könnte Bilderkennungssoftware Ärzte bei der Diagnose von Krebs oder Knochenbrüchen unterstützen.
„KI-Methoden könnten aber auch dazu beitragen, schneller einen Wirkstoff gegen das neue Coronavirus SARS-CoV-2 zu finden“, sagt Prof. Dr. Oliver Kramer. Der Informatiker leitet die Arbeitsgruppe Computational Intelligence an der Universität Oldenburg und beschäftigt sich mit KI-Verfahren wie Deep Learning oder evolutionären Algorithmen. Das gezielte Design von Wirkstoffen, also das Entwerfen passender chemischer Verbindungen für einen bestimmten Zweck, zählte bislang nicht zu seinen Schwerpunkten. Doch als sich die Corona-Epidemie Anfang des Jahres auszubreiten begann, beschloss Kramer, sich in das Gebiet einzuarbeiten – auch, um zu zeigen, dass die KI-Forschung neben einigen oft kritisch diskutierten Anwendungen dazu beitragen kann, drängende gesellschaftliche Probleme zu lösen.
Aufwändige Medikamentensuche
Die Suche nach einem passenden Wirkstoff für eine Krankheit ist derzeit ein aufwändiger Prozess, der mit Labortests beginnt und mit klinischen Studien endet. In der Regel zieht er sich über mehrere Jahre hin. Um geeignete Substanzen zu finden, testen Pharmakologen zunächst Tausende von möglichen Wirkstoffen mit vollautomatischen Verfahren auf ihre biologische Wirkung. Parallel kommen Computerprogramme zum Einsatz, die Substanzen mit möglichst passenden physikalischen Eigenschaften gezielt entwerfen, etwa, um eine ganz bestimmte biochemische Reaktion in einer Zelle zu blockieren. KI-Verfahren könnten diese Suche beschleunigen, beispielsweise durch selbstlernende Algorithmen, die mit der Zeit immer bessere Ergebnisse liefern. Derzeit stehen diese Ansätze allerdings noch am Anfang.
Kramer hat nun gemeinsam mit fünf Mitarbeitern ein KI-Verfahren entwickelt, um Substanzen aufzuspüren, die ein bestimmtes Enzym des neuen Coronavirus SARS-CoV-2 lahmlegen könnten. Den Informatikern geht es dabei zunächst weniger darum, tatsächlich ein potentielles Medikament gegen COVID-19 zu entwickeln. „Unser Beitrag ist vor allem ein methodischer Vorschlag, bei dem wir unsere KI-Kompetenzen einbringen können“, betont Kramer.
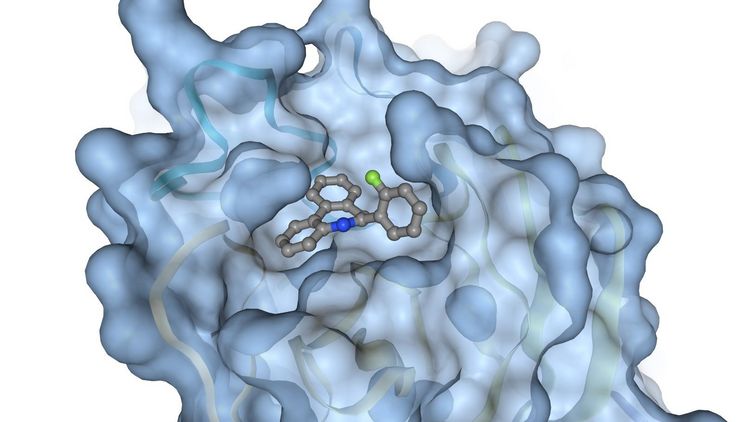
Das Team, zu dem auch Thomas Teusch aus der Arbeitsgruppe Theoretische Chemie zählte, wählte als Ziel die so genannte Hauptprotease des Virus. Der Erreger braucht dieses Enzym als Werkzeug, um sich zu vermehren. Die Protease ist ein großes und komplexes Biomolekül, das lange Eiweißketten in kleinere Schnipsel zerschneidet. Nachdem chinesische Forscherinnen und Forscher bereits im Januar das Genom und damit den Bauplan von SARS-CoV-2 entschlüsselt hatten, gelang es einem Team der Universität Lübeck kurz darauf, die kristalline Struktur der Protease aufzuklären und damit die Voraussetzung für die Suche nach einem Medikament zu schaffen. Kramer erläutert: „Die Idee besteht darin, ein Molekül zu finden, das sich an einer bestimmten Stelle der Coronavirus-Protease anheftet und so ihre Funktion stört.“ Ein solcher Wirkstoff könnte den Lebenszyklus des Virus unterbrechen und somit den Krankheitsverlauf abmildern.
Tests mit dem Computer
Wie bei der computerbasierten Wirkstoffsuche üblich, repräsentieren die Informatiker in ihrem Modell chemische Verbindungen durch Zeichencodes, die jedes einzelne Atom und seine Lage im Molekül jeweils exakt beschreiben. Anhand von bestimmten Kriterien testet ihr Programm anschließend, wie gut sich eine Substanz als Wirkstoff eignet. Dabei nutzen die Forscher ein evolutionäres Verfahren: Beginnend mit zehn oder 20 Ausgangsstoffen erzeugen sie durch zufällige Veränderungen des Zeichencodes neue Verbindungen, die das Programm ebenfalls auf ihre Eignung als Wirkstoff testet. Aus jeder so entstandenen Generation wählt das Programm die Verbindungen mit den besten Eigenschaften aus, um daraus neue Tochter-Substanzen zu erzeugen. Nach diesem Prinzip ließen die Forscher das Programm mit unterschiedlichen Einstellungen mehrfach so lange laufen, bis sich diese Abfolge 200-mal wiederholt hatte. Anschließend analysierten sie, wie gut die Substanzen, die am Ende herauskamen, die gewünschten Kriterien erfüllten.
„Unser Ansatz gehört zu den wenigen, die mehrere Eigenschaften von Molekülen gleichzeitig optimieren und nicht nur eine einzige“, sagt Kramer. Die gefundenen Wirkstoff-Kandidaten wiesen typische Merkmale von Medikamenten auf: Viele enthielten Benzolringe, also Strukturen aus sechs Kohlenstoff-Atomen, außerdem waren die meisten Moleküle eher klein, was für wirksame Protease-Inhibitoren nicht untypisch ist. Einige Substanzen, die besonders gut an die SARS-CoV-2-Protease andocken konnten, wiesen eine ungewöhnliche Geometrie auf.
Diese Erkenntnisse könnten zumindest den Startpunkt für weitere Untersuchungen bilden, sagt Kramer. Um realistischere Ergebnisse zu erzielen, muss seiner Meinung nach unter anderem noch das physikalische Modell verbessert werden, das berechnet, wie fest sich mögliche Wirkstoffe mit der Protease verbinden. Die Forscher verwendeten dafür ein gängiges Programm, das zahlreiche Vereinfachungen enthält, um die Rechenzeit zu verkürzen.
Bei der Veröffentlichung ihrer Ergebnisse haben Kramer und seine Kollegen einen immer beliebteren Weg gewählt: Sie stellten die Untersuchung auf einem so genannten Preprint-Server vor und baten unterschiedliche Experten um Feedback. Gleichzeitig meldeten Sie den Artikel als Beitrag bei einer wichtigen Konferenz an, wo er inzwischen begutachtet und akzeptiert wurde. „Uns ist es wichtig, dass der Ansatz nicht in der Schublade verschwindet. Wir möchten unsere Arbeit der Fachwelt zugänglich machen, neue Impulse aufnehmen und dann weiter daran forschen“, betont der Informatiker. Aus diesem Grund habe sich das Team entschieden, frühzeitig an die Öffentlichkeit zu gehen. Bislang seien die Rückmeldungen durchweg positiv: „Die Kritikpunkte, die wir erhalten haben, gingen bereits sehr stark ins Detail“, so Kramer. „Das lässt uns hoffen, dass wir auf dem richtigen Weg sind.“